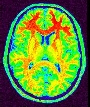





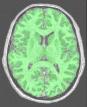
Génère un masque binaire du cerveau à partir de l'IRM brute
Cette procédure s'appuie sur le fait que le cerveau est un objet clair et massif entouré d'un liseré noir correspondant au crâne, qui l'isole en grande partie du reste des tissus. La procédure d'analyse d'images cherche à casser les quelques connections du cerveau avec le monde extérieur à partir d'un processus d'érosion très similaire aux phénomènes géologiques ou à l'action de la rouille lorsqu'elle fragilise les objets métalliques au niveau de leurs goulots d'étranglement avant de les casser.
L'image en niveaux de gris est dans un premier temps seuillée de manière à obtenir un objet binaire (blanc sur un fond noir). Dans un second temps, un processus d'érosion est simulé de manière à rogner cet objet binaire sur une certaine épaisseur. Cette érosion engendre plusieurs morceaux déconnectés. Le plus gros d'entre eux est sélectionné en tant que graine du cerveau. Un processus de croissance permet alors de faire croître cette graine de manière à récupérer la forme du cerveau.
Une illustration du processus au niveau d'une coupe:
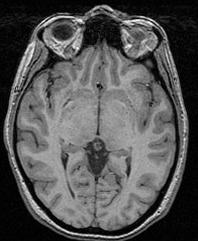
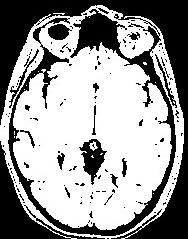
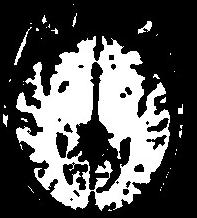

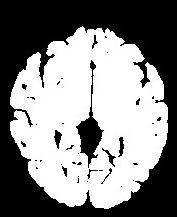
Pour parvenir à ses fins, cette moulinette enchaîne 3 étapes:La procédure réalisant la segmentation intègre de nombreuses "verrues" visant à résoudre certaines difficultés apparaissant parfois pour certaines séquences ou certaines morphologies. Si vous observez son défilement dans la fenêtre de log, vous aurez un apercu d'une partie de ces ajouts. Le plus important est le réglage automatique de la taille d'érosion optimale pour une image donnée.
- Une correction du biais spatial (Vip Bias Correction). Il s'agit de l'étape clef de tout le processus. Si elle ne se passe pas parfaitement, les résultats en patiront. Il est donc important de s'assurer qu'elle se comporte bien sur quelques images de votre scanner avant de chercher à aller plus loin. Les deux choix proposés au départ (contrast, type de biais) sont dédiés à cette procédure.
- Une analyse de l'histogramme (Vip Histogram Analysis)
- Une segmentation du cerveau (Vip Get Brain)
Si le fichier indiquant la localisation des commissures a été spécifié, une vérification finale tentera d'éliminer d'éventuels résidus des sinus/méninges faisant le tour du cerveau au niveau du plan interhémisphérique.
Si un masque de lésion est spécifié, la région indiquée sera ajoutée à la binarisation initiale quelque soient les niveaux de gris à l'intérieur. Cela corrigera les situations où, du fait de la présence d'une grosse lésion, une partie du cerveau disparaît au cours du processus d'érosion/dilatation (lorsque la graine est segmentée en plusieurs morceaux).
Vérification du résultat et corrections éventuelles:
Cf. outils brainvisa:
Validation Brain Mask from T1 MRI
Correction Brain Mask from T1 MRI
La première manière de procéder consiste à visualiser simultanément l'image corrigée du biais et le masque du cerveau, en s'aidant du curseur lié d'Anatomist :
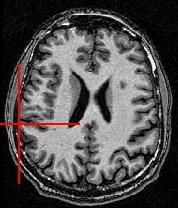
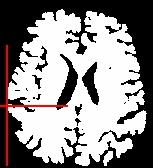
Une seconde manière de procéder consiste à fusionner les deux images. L'oeuil de brainVISA le fait automatiquement. Manuellement, il vous faut fusionnez les deux volumes, appliquez une table uni-couleur dotée de l'extension ufusion au masque, puis passez la fusion en mode combinaison linéaire:

Si tout vous semble correct, vous pouvez passer à une autre étape...
Vous pouvez d'ailleurs signaler à brainvisa de ne plus remettre en question ce résultat avec la procédure: Validation Brain Mask from T1 MRI
Dans le cas contraire, il va vous falloir exécuter la procédure d'erreur associée à cette moulinette:
Correction Brain Mask from T1 MRI
T1mri: Raw T1 MRI ( entrée )
Contrast: Choice ( input )
Bias_type: Choice ( input )
mri_corrected: IRM T1 Biais Corrigé ( sortie )
histo_analysis: Analyse d'histogramme ( sortie )
brain_mask: T1 Brain Mask ( sortie )
Commissure_coordinates: Commissure coordinates ( optional, entrée )
lesion_mask: Volume 3D ( optional, entrée )masque binaire d'une lésion (optionnel)
Toolbox : Morphologist
Niveau d'utilisateur : 2
Identifiant :
AnaT1toBrainMaskNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/segmentation/AnaT1toBrainMask.pySupported file formats :
T1mri :GIS image, VIDA image, NIFTI-1 image, MINC image, gz compressed MINC image, DICOM image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, TIFF(.tif) image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imagemri_corrected :GIS image, VIDA image, NIFTI-1 image, MINC image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imagehisto_analysis :Analyse d'histogrammebrain_mask :GIS image, VIDA image, NIFTI-1 image, MINC image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imageCommissure_coordinates :Commissure coordinateslesion_mask :GIS image, VIDA image, NIFTI-1 image, MINC image, gz compressed MINC image, DICOM image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, TIFF(.tif) image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v image