

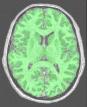




Segmente le masque du cerveau en trois parties : les hémisphères cérébraux et le reste (cervelet/tronc) (procédure fondée sur Vip Split Brain)
Cette procédure vise à séparer les hémisphères et à éliminer le cervelet et une partie du tronc cérébral de manière à accéder aux faces internes et basses du cortex. Elle s'appuie sur un principe similaire à celui utilisé pour segmenter le cerveau (Brain Mask From T1 MRI). Une érosion est appliquée à un masque de la matière blanche afin de la casser au niveau du corps calleux et du tronc cérébral. On récupère alors 3 graines correspondant aux hémisphères et au cervelet. Ces graines sont alors amenées à croître, dans un premier temps au sein de la matière blanche, puis au sein de la matière grise, de manière à retrouver les formes des hémisphères.
L'image résultat s'appelle un diagramme de Voronoï, c'est-à-dire une parcellisation d'un domaine en fonction de la graine la plus proche. Ce diagramme ressemble aux zones d'influence des chateaux du moyen âge, dont le rôle est ici joué par les graines.
Une illustration du principe du processus appliqué ici avec cinq graines:
Un premier diagramme de Voronoï est construit à partir des 5 plus grosses graines issues d'une érosion de la matière blanche:
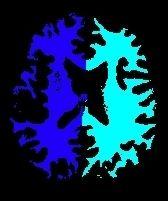

Ce premier diagramme fournit les graines permettant d'en construire un second au sein de l'ensemble du cerveau:


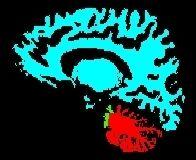
Si la localisation des commissures est spécifiée, un prédécoupage sera effectué dans la région du corps calleux et du tronc cérébral pour simplifier la séparation des graines.
Si le template de diagramme de Voronoï est également spécifié, il permettra à la procédure de déterminer la taille d'érosion qui permet d'obtenir les graines recherchées. Une région donnée (un hémisphère ou le cervelet) pourra alors être issue d'une graine dotée de plusieurs composantes connexes, ce qui augmente largement la robustesse de la procédure. En effet le processus d'érosion peut facilement séparer les hémisphères du cervelet, ou séparer le lobe temporal du reste du cerveau. Dans ces cas critiques, si les objets ne sont représentés que par une seule composante connexe, les frontières du diagramme de Voronoï final peuvent être mal placées.
Lorsque ces deux options sont spécifiées, la principale faiblesse de la procédure se situe au niveau de l'interface hémisphères/cervelet. Il s'agit en effet d'une interface gris/gris parfois difficile à déterminer du fait de l'effet de volume partiel mélangeant matière grise et matière blanche dans le cervelet.Vérification du résultat et corrections éventuelles:
Cf. outils brainvisa:
Validation Split Brain from Brain Mask
Correction Split Brain from Brain Mask
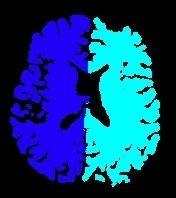
Le résultat final, que vous pouvez visualiser en fusionnant l'image IRM et le diagramme de Voronoï, doit ressembler à ce qui suit :

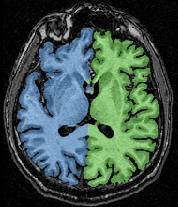
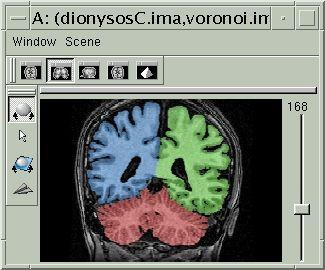


Si tout vous semble correct, vous pouvez passer à une autre étape...
Vous pouvez d'ailleurs signaler à brainvisa de ne plus remettre en question ce résultat avec la procédure:Validation Split Brain from Brain Mask
Dans le cas contraire, il va vous falloir exécuter la procédure d'erreur associée à cette moulinette (à venir):
Correction Split Brain from Brain Mask
mri_corrected: IRM T1 Biais Corrigé ( entrée )
histo_analysis: Analyse d'histogramme ( entrée )
brain_mask: T1 Brain Mask ( entrée )
Use_template: Booléen ( input )Utilisation du modèle
split_template: Hemispheres Template ( optional, entrée )
split_mask: Séparation du masque du cerveau ( sortie )
Commissure_coordinates: Commissure coordinates ( optional, entrée )pour l'utilisation du repère de Talairach
Toolbox : Morphologist
Niveau d'utilisateur : 2
Identifiant :
AnaSplitBrainFromBrainMaskNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/segmentation/AnaSplitBrainFromBrainMask.pySupported file formats :
mri_corrected :GIS image, VIDA image, NIFTI-1 image, MINC image, gz compressed MINC image, DICOM image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, TIFF(.tif) image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imagehisto_analysis :Analyse d'histogrammebrain_mask :GIS image, VIDA image, NIFTI-1 image, MINC image, gz compressed MINC image, DICOM image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, TIFF(.tif) image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imagesplit_template :GIS image, VIDA image, NIFTI-1 image, MINC image, gz compressed MINC image, DICOM image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, TIFF(.tif) image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imagesplit_mask :GIS image, VIDA image, NIFTI-1 image, MINC image, TIFF image, XBM image, PBM image, PGM image, BMP image, XPM image, PPM image, gz compressed NIFTI-1 image, ECAT i image, PNG image, JPEG image, MNG image, GIF image, SPM image, ECAT v imageCommissure_coordinates :Commissure coordinates